Erste 3D-Bilder eines gigantischen Moleküls
24.03.2021Die Struktur großer, in ihrer Form variabler Molekülkomplexe zu erfassen, ist extrem schwierig. Wissenschaftlern aus Würzburg und Montpellier ist dies jetzt dank eines neuen Ansatzes bei einer wichtigen Proteinmaschine gelungen.
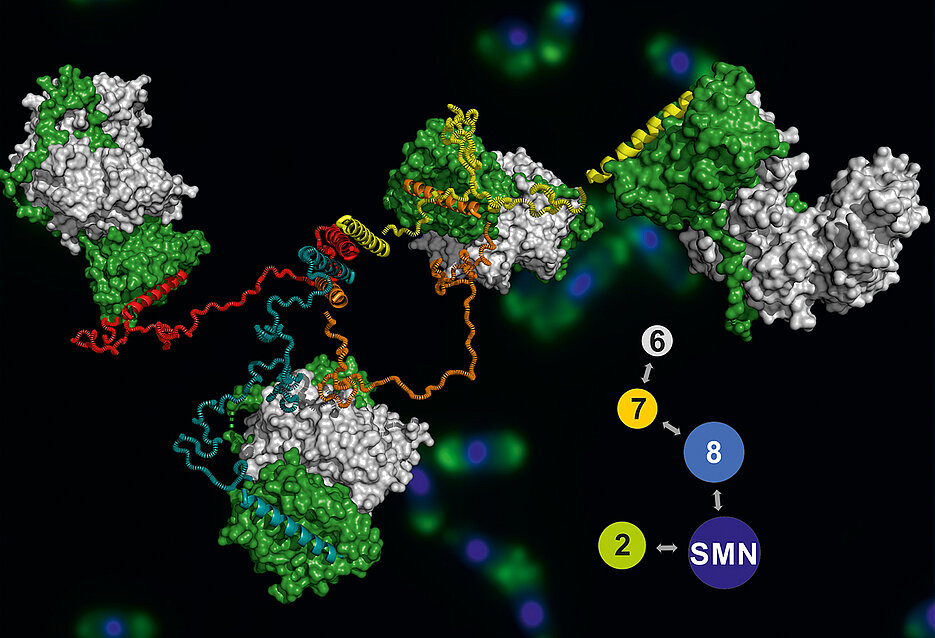
SMN oder ausgeschrieben Survival Motor Neuron: Schon seit vielen Jahren forscht Professor Utz Fischer an diesem Protein und dem großen gleichnamigen Molekülkomplex, zu dessen Bausteinen auch SMN gehört. Fischer ist Inhaber des Lehrstuhls für Biochemie an der Julius-Maximilians-Universität Würzburg (JMU). Auf das Molekül gestoßen ist er bei seiner Suche nach dem Auslöser der Spinalen Muskelatrophie. Diese Krankheit wird, wie sich vor einigen Jahren herausstellte, durch einen Mangel an SMN hervorgerufen.
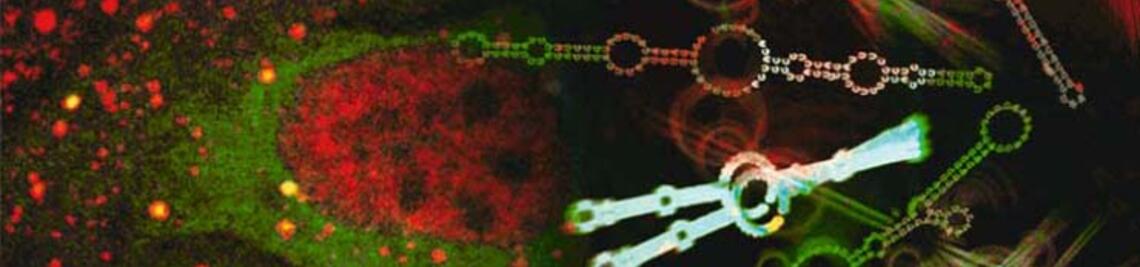
Jetzt ist es dem Arbeitskreis um Fischer gelungen, ein erstes dreidimensionales Modell des gesamten SMN-Komplexes zu präsentieren. Mit dem Wissen über seine Struktur ist es möglich zu verstehen, wie der Komplex arbeitet und warum sein Funktionsverlust zur Muskelatrophie führt. Die Ergebnisse ihrer Arbeit haben die Wissenschaftler in der aktuellen Ausgabe der Fachzeitschrift Nucleic Acids Research veröffentlicht. Für das Journal handelt es sich um einen „Breakthrough Article“.
Möglich gemacht hat die neuen Erkenntnisse ein integrativer strukturbiologischer Ansatz, bei dem biochemische, genetische und biophysikalische Techniken miteinander kombiniert wurden.
Auflösung bis zum millionsten Teil eines Millimeters
„Die Auflösungsrevolution, die primär durch die Entwicklung in der Cryo-Elektronenmikroskopie ermöglicht wurde, hat die Strukturanalyse von großen und komplexen zellulären Molekülen und Molekülverbänden mit atomarer Detailtreue ermöglicht“, sagt Utz Fischer. Der einzige Haken an der Technik ist jedoch, dass sie vor allem bei solchen Strukturen sehr gut funktioniert, die weitgehend starr sind und wenige flexible Bereiche aufweisen.
Leider ist dies bei vielen Molekülverbänden nicht der Fall, so auch beim SMN-Komplex. „Dieser Komplex ist zentral für unsere Zellen, da er beim Aufbau von wichtigen molekularen Maschinen hilft“, sagt Fischer. Dafür muss er allerdings sehr flexibel und dynamisch sein, um seine Funktion in Zellen erfüllen zu können. Das verhinderte seine Strukturanalyse über althergebrachte Strategien bislang.
Eine Kombination unterschiedlicher Methoden führt zum Ziel
Fischer und sein Team haben deshalb einen alternativen Weg gewählt: „Ausgangspunkt war eine Kooperation mit der Gruppe von Dr. Rémy Bordonné aus dem französischen Montpellier, durch die der SMN-Komplex aus der Hefe Schizosaccharomyces pombe identifiziert werden konnte“, erklärt der Biochemiker. Da dieser aus weniger Einzelkomponenten besteht als der menschliche Komplex und sich auch weniger dynamisch verhält, war er für eine integrative strukturbiologische Untersuchung ideal.
„Wir haben zunächst einzelne Teilbereiche, die für den Zusammenhalt des Komplexes wichtig sind, mit Hilfe der Röntgenstrukturanalyse sichtbar gemacht“, beschreibt Fischer das Vorgehen der Wissenschaftler. In einem zweiten Schritt haben sie dann den Gesamtkomplex und Teile davon mittels der Röntgen-Kleinwinkelstreuung charakterisiert. Diese Methode liefert auch Informationen über das dynamische Verhalten ungefalteter Bereiche des Komplexes. Anschließend wurden fehlende Bereiche mit dem bioinformatischen Verfahren der 3D-Homologiemodellierung rekonstruiert.
Diese Kombination unterschiedlicher strukturbiologischer Methoden wird wegen ihrer bislang unerreichten Resultate in Zukunft an Bedeutung gewinnen, ist sich Dr. Clemens Grimm sicher. Er leitet an Fischers Lehrstuhl den Bereich „Strukturanalyse“ und war ebenfalls an der jetzt veröffentlichten Arbeit beteiligt.
Ein Oktopus mit flexiblen Armen
Das Ergebnis war ein Modell des gesamten SMN-Komplexes, das dessen Funktion sehr gut erklärt: Ähnlich wie bei einem Oktopus erstrecken sich von einem zentralen „Körper“ des Komplexes mehrere lange und sehr flexible „Arme“. Über diese Arme kann der Komplex Proteine einfangen und zusammen mit anderen Biomolekülen zu molekularen Maschinen zusammenfügen.
Das Modell liefert damit auch neue Einblicke in die Entstehungsprozesse der Spinalen Muskelatrophie. „Mutationen, welche diese Krankheit verursachen, liegen gehäuft im zentralen Körper“, sagt der Fischers Doktorand Jyotishman Veepaschit, der diese Experimente gemeinsam mit seinem Kollegen Aravindan Viswanathan durchgeführt hat. Sie verhindern, dass sich der Komplex vollständig ausbilden und seine Funktion in der Zelle wahrnehmen kann.
Originalpublikation
Identification and structural analysis of the Schizosaccharomyces pombe SMN complex. Jyotishman Veepaschit, Aravindan Viswanathan, Rémy Bordonné, Clemens Grimm, and Utz Fischer. Nucleic Acids Research. doi: 10.1093/nar/gkab158
Stichwort Spinale Muskelatrophie
Die Spinale Muskelatrophie ist eine erbliche Krankheit, die im Kinder-, Jugend- oder Erwachsenenalter auftreten und unterschiedlich schwer ausgeprägt sein kann. Bei besonders schlimmen Formen sterben die Betroffenen an den Folgen einer Muskelschwäche und fortschreitenden Lähmungen schon im Säuglingsalter. In anderen Fällen können die Ärzte durch Krankengymnastik und orthopädische Hilfsmittel die Mobilität und Vitalität für längere Zeit erhalten.
Die Spinale Muskelatrophie ist nicht selten: Sie trifft etwa einen von 6.000 Menschen. Auf Grund einer Veränderung des Erbguts besitzen die Betroffenen zu wenig von dem Protein SMN. Ein Mangel, der sich besonders in denjenigen Nervenzellen im Rückenmark zeigt, die die Bewegung der Muskeln steuern: Diese so genannten Motoneuronen verlieren den Kontakt zum Muskel und sterben ab.
Kontakt
Prof. Dr. Utz Fischer, Lehrstuhl für Biochemie, T: +49 931 31-84029, utz.fischer@biozentrum.uni-wuerzburg.de