Prof. Dr. Dr. Martin J. Müller
Lehrstuhl für Pharmazeutische Biologie
Julius-von-Sachs-Institut für Biowissenschaften
Julius-von-Sachs-Platz 2
97082 Würzburg
Tel.: + 49 931 31-86160
Fax: + 49 931 31-86182
martin.mueller@uni-wuerzburg.de
CV
Publikationen
Schwerpunkte der Arbeitsgruppe
Die Arbeitsgruppe untersucht Lipidsignalwege sowie den Metabolismus von Lipiden und Sekundärstoffen in pflanzlichen Anpassungsreaktionen auf biotischen und abiotischen Stress. Massenspektrometrie-basierte metabolische Analysen werden auch am Modellorganismus Drosophila durchgeführt, um den Einfluss der inneren Uhr auf die Steuerung des Metabolismus aufzuklären.
Wir setzen Metabolomics/Lipidomics meist als ersten Forschungsansatz ein, um schnelle metabolische Adaptationsmechanismen erkennen zu können. Die Umsteuerung des Metabolismus an eine veränderte Umgebung ist eine essentielle und unmittelbare Anpassungsreaktion, die im Falle des Versagens erhebliche Fitnesskonsequenzen haben kann. Wir nutzen genetische Methoden, um die Relevanz der metabolischen Feinsteuerung funktionell im Kontext von biotischem und abiotischen Stress zu testen.
Lipidmediatoren in Pflanzen
Martin J. Müller (PI)
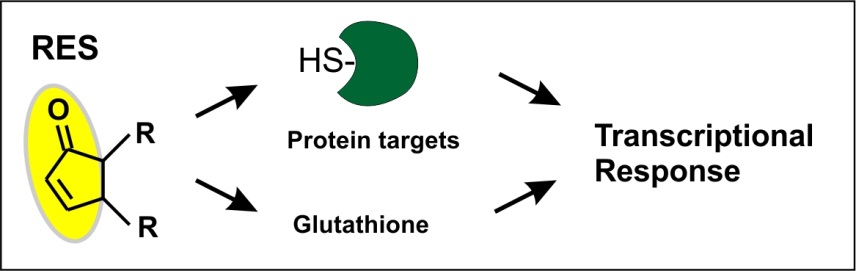
Oxidierte Lipide in Mikroben, Pflanzen und Tieren spielen oft eine essentielle Rolle als Signalstoffe in der Entwicklung, der Reproduktion sowie der Abwehr von biotischem und abiotischem Stress. Neben Jasmonaten fokussieren wir auf neue, elektrophile Lipidmediatoren sowie Sekundärstoffe, die Abwehrreaktionen durch kovalente Modifikation von Proteinen und Änderungen des zellulären Redoxpotentials vermitteln. Die rasche Aktivierung Lipid-basierter Abwehrmechanismen ist eine wichtige ‚first-line‘ Abwehrstrategie gegen Mikroben und Herbivore. Ziel des Projektes ist die Aufklärung der zugrundeliegenden Erkennungs- und Signaltransduktionsmechanismen sowie der Bedeutung der Signalstoffe für spezifische Abwehrreaktionen.
Metabolische Mechanismen der Thermotoleranz in Pflanzen
Martin J. Müller (PI), Agnes Fekete (PI)
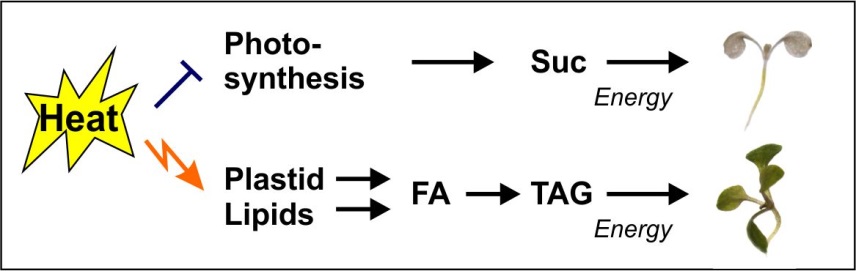
In Vorarbeiten am Lehrstuhl konnte gezeigt werden, dass Pflanzen schon bei moderatem Hitzestress Strukturlipide aus Membranen in Energiespeicherlipide (Triacylglyceride) umwandeln. Mutanten mit einem Defekt in der metabolischen Adaptation an Hitze zeigten sich als hochgradig sensitiv gegenüber Hitzestress. Eigene und kürzlich von anderen Arbeitsgruppen publizierte Arbeiten deuten darauf hin, dass der schnelle, stress-induzierte Aufbau von Energiespeicherlipiden in vegetativen Pflanzengeweben eine wichtige Funktion bei der Resistenz gegenüber abiotischem und biotischem Stress, der mit einer Beeinträchtigung der Energiebereitstellung durch Photosynthese einhergeht, hat. Die metabolischen Anpassungsreaktionen von Pflanzen an Hitzestress (und andere Stressoren) werden im Detail aufgeklärt und deren funktionelle Bedeutung mittels genetischer, physiologischer und metabolmomischer Methoden aufgeklärt.
Pflanzliche Thermotoleranz - Genome-wide association studies
Daniel Maag (PI), Martin J. Müller, Arthur Korte (CCTB)
Arabidopsis thaliana ist auf der gesamten Nordhalbkugel verbreitet. Dabei haben sich Ökotypen herausgebildet, die an klimatisch völlig verschiedene Umweltbedingungen angepasst sind. Wir nutzen eine Kollektion von über 1135 verschiedenen Ökotypen mit sequenzierten Genom, um spezielle Adaptationen der Thermotoleranz an das jeweilige Klima mit genetischen Veränderungen zu korrelieren. Ziel ist es, Gene zu identifizieren und funktionell zu validieren, die mit der Hitze-Wahrnehmung und Signalweiterleitung assoziiert sind. Dabei kommen genetische, metabolische, physiologische und bioinformatorische Methoden zur Anwendung.
"Insekt timing": Einfluss der inneren Uhr von Drosophila auf die Steuerung des Metabolismus
Agnes Fekete (PI), Martin J. Mueller (PI)
Korrektes ‘Timing’ ist essentiell für alle Organismen. In dem SFB "Insect timing"- Project verwenden wir einen high-end massenspektrometrischen Metabolomics Ansatz, um Uhr-gesteuerte, circadian regulierte Metabolite in Drosophila melanogaster zu identifizieren. Diese Analysen werden ergänzt durch molekulare und physiologische Untersuchen in enger Zusammenarbeit mit den anderen SFB-Teilprojekten. Ziel des Projektes ist es den funktionellen Zusammenhang von Uhrgenen mit dem Metabolismus und der Physiologie zu verstehen.