Forschung Arbeitsgruppe Dröge-Laser
Bereich A: Energiehomöostase
Bereich B: Arabidopsis Transkriptionsfaktoren
Signaltransduktionsprozesse der pflanzlichen Antwort auf Energiemangel
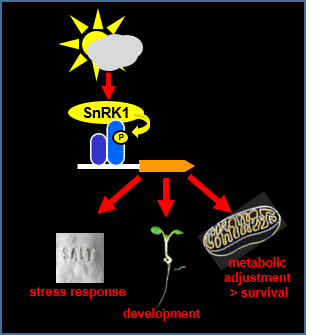
Die Kontrolle der Energie-Homeostase ist von entscheidender Bedeutung für alle Lebewesen. Pflanzen besitzen ein evolutionär konserviertes System bestehend aus zwei antagonistisch wirkender Kinasen: TOR (TARGET OF RAPAMYCIN) fördert anabolische, energieverbrauchende Prozesse, während unter Energiemangel SnRK1 (Snf1 RELATED KINASE 1) katabolische, energiebereitstellende Prozesse aktiviert. Unter natürlichen Bedingungen führen Schwachlicht oder biotischer und abiotischer Stress zu Energiemangelsituationen. In diesem Projekt konnten wir basic leucine Zipper (bZIP) Transkriptionsfaktoren der Gruppe C und S1 als Zielproteine und Mediatoren der SnRK1-Antwort identifizieren. Wir befassen uns (1) mit den Mechanismen wie Energiemangelsignale mittels SnRK1 und C/S1-bZIP-Faktoren die Transkription kontrollieren und (2) welche Funktionen durch die SnRK1-C/S1-bZIP-Signaltransduktion reguliert werden. Hier analysieren wir alternative metabolische Wege zum Abbau von Aminosäuren zum Erhalt der mitochondrialen ATP Produktion unter Stress, energieabhängige Entwicklungsprozesse wie Wurzelwachstum, Keimung, Dunkel-induzierte sowie Salzstress oder Pathogenantwort.
Literatur: Muralidhara et al PNAS 2021; Henniger et al. Plant Cell 2021; Pedrotti et al., Plant Cell 2018, Dröge-Laser and Weiste TIPS 2018, Nukarinen et al., Sci. Rep. 2016, Mair et al., eLife 2015, Hartmann et al., Plant Cell 2015, Dietrich et al., Plant Cell 2011 .
Mitarbeiter: Christoph Weiste, Jan Draken, Monika Kumari, Theresa WIldenhain
Anpassung Auxin-vermittelter Wachstumsprozesse an die zellulären Energiereserven
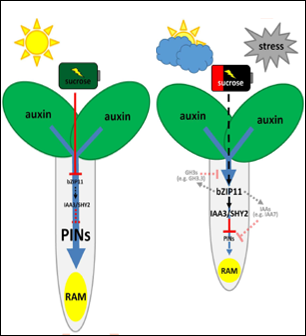
Das Pflanzenhormon Auxin kontrolliert vielfältige Wachstums- und Entwicklungsantworten, die i. W. durch Genexpression vermittelt werden. Transkriptionsfaktoren der ARF-Familie (Auxin Response FactorS) sind hier von entscheidende Bedeutung. Pflanzliches Wachstum ist begrenzt, z. B. durch Verfügbarkeit von Energie, C- und N-Ressourcen. Wir verfolgen die Hypothese, dass C/S1-bZIP-Faktoren, durch Interaktion mit ARFs, Auxin-vermittelte Genexpression bzw. Wachstumsprozesse an die verfügbare zelluläre Energiereserven anpassen.
Literatur: Weiste et al., PLoS Genetics 2017; Weiste und Dröge-Laser, Nature Com. 2014
Mitarbeiter: Christoph Weiste
Methoden zur Analyse des Transkriptionsfaktor ORFeoms in Arabidopsis
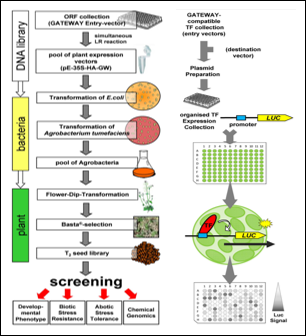
Transkriptionsfaktors (TFs) sind wichtige zelluläre Regulatoren der Transkription. In der Modelpflanze Arabidopsis thaliana sind mehr als 2000 TF identifiziert und stehen als Open Reading Frame (ORFs) Kollektionen zur Verfügung. Hier beschäftigen wir uns mit der Etablierung und Nutzung molekularer Methoden zur funktionalen Charakterisierung dieses “ORFeomes”.
> Arabidopsis thaliana Transcription Factor Open Reading Frame Expression
(AtTORF-Ex)
Herstellung und „Screening“ einer Kollektion von TF-Überexpressionslinien (AtTORF-Ex) mit dem Ziel, Pflanzen mit erhöhter Stresstoleranz zu isolieren.
> Protoplast Trans Activation (PTA) Screening
Zur Identifizierung von TF als Regulatoren eines Promotors der Wahl wurde ein hoch-durchsatz Protoplasten Transformationssystem entwickelt. Nach Co-expression eines Promotor:LUCIFERASE-Konstrukts und einer Kollektion von z. Zt. ca. 1700 GATEWAY-kompatiblen TF-Expressionsvektoren können transkriptionelle Aktivatoren und Repressoren identifiziert werden.
Literatur: Dröge-Laser et al. COPS 2018, Wehner et al. 2011a, b; Zhang et al., 2015 Weiste et al., 2007
Mitarbeiter: Monika Kumari, Jan Draken, Christoph Weiste
Bereich C: Pflanzen-Pathogen-Interaktion
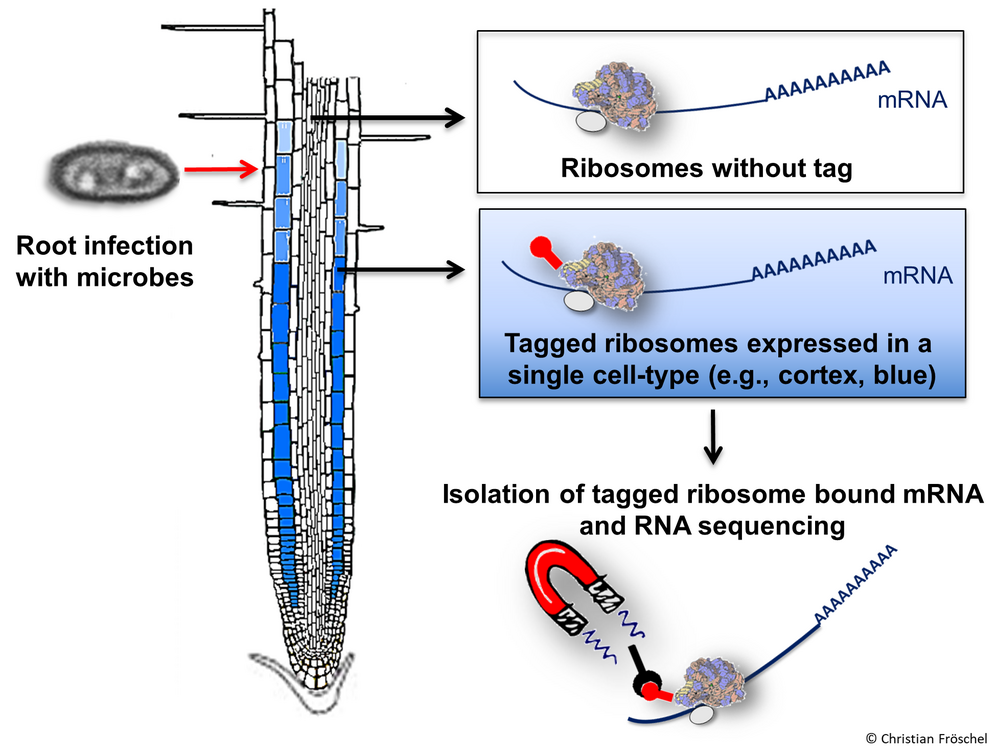
Genomweite Analyse der Zellschicht-spezifischen Expression Pathogen-induzierter Gene in der Arabidopsis Wurzel
Obwohl Pflanzenwurzeln mit einer Vielzahl von Mikroorganismen in engem Kontakt stehen, sind induzierbare Abwehrreaktionen der Wurzel gegen Pathogene bisher wenig beschrieben. Der durch konzentrischen Zellschichten charakterisierte Wurzelaufbau lässt vermuten, dass diese Schichten durch spezifische Programme einen Beitrag zur Pathogenabwehr leisten. In diesem Projekt wurde für die Modellpflanze Arabidopsis eine iTRAPseq Methodik (infektionsgekoppelte Translating Ribosome Affinity Purification und anschließende RNA-Sequenzierung) etabliert, um zellschichtspezifische mRNA zu isolieren und analysieren. Damit konnten wir zeigen, dass die Wurzelzellschichten auf mikrobielle Infektionen mit spezifischen, genetisch definierten Programmen reagieren (Fröschel et al., 2021). Zellschichtspezifische Antworten auf Infektionen mit pathogenen und mutualistischen Pilzen werden verglichen und ihre Bedeutung für die Wurzel-Mikroorganismus Interaktionen wird untersucht.
Literatur: Fröschel and Dröge-Laser Mol. Plant, 2023; Fröschel et al., Cell Host and Microbe, 2021
Mitarbeiter: Alexander Marsell
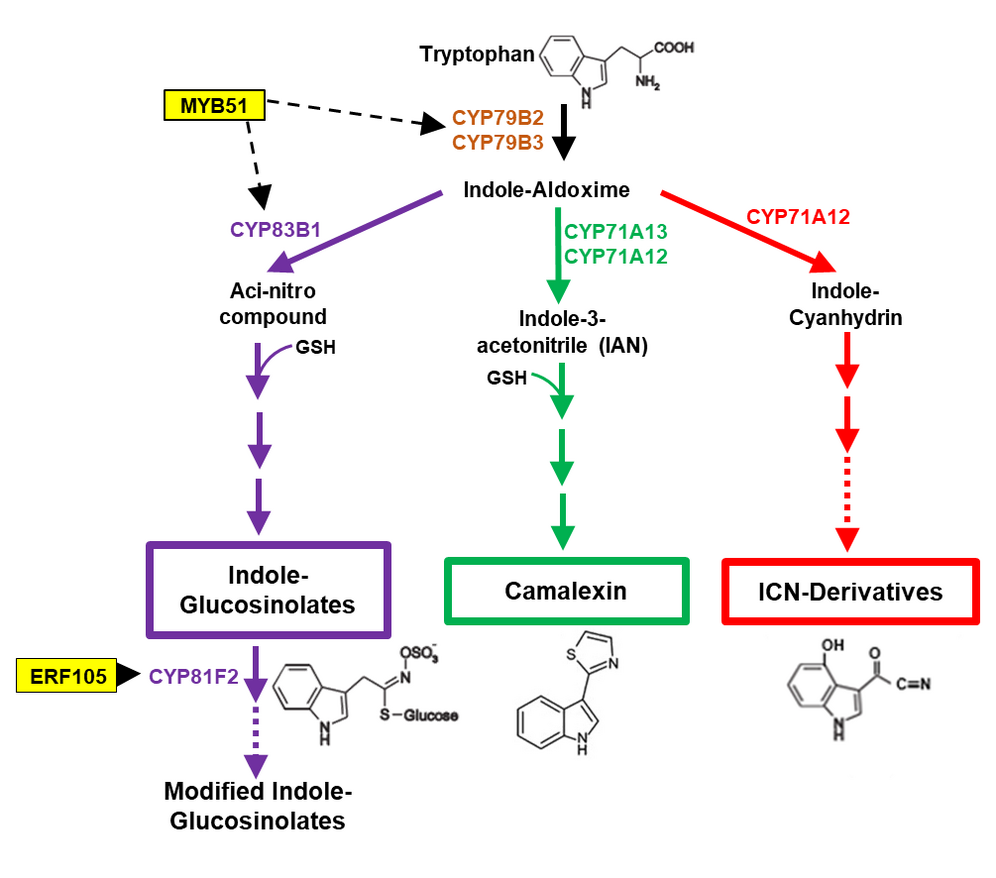
Transkriptionsfaktor-Netzwerk zur Kontrolle der Biosynthese Tryptophan-abgeleiteter antimikrobieller Verbindungen
Die Biosynthese von Sekundärmetaboliten mit antimikrobieller Aktivität ist eine effektive Strategie in Pflanzen, um Mikroorganismen abzuwehren. In der Modellpflanze Arabidopsis leiten sich die antimikrobiellen Verbindungen Camalexin, Indol-Glucosinolate (IG) sowie ICN-Derivate von der Aminosäure Tryptophan (Trp) ab. Der komplexe Trp-abhängige Sekundärmetabolismus wird auf Transkriptionsebene sehr koordiniert reguliert und stellt somit ein ideales Modellsystem für die Analyse regulativer Transkriptions-Netzwerke dar. Wir konnten mit Hilfe von „Screening“-Ansätzen wichtige Regulatoren des IG Zweiges identifizieren, etwa einige Mitglieder der ERF (Ethylen Response Factor) und MYB (MYB Domain Protein) Transkriptionsfaktorfamilien. Ziel dieses Projektes ist es, weitere „Master“-Regulatoren im Trp-abhängigen Sekundärmetabolismus zu identifizieren.
Literatur: Iven et al., Mol. Plant 2012; Fröschel et al., MPMI 2019; Fröschel et al., Cell Host and Microbe 2021
Mitarbeiter:
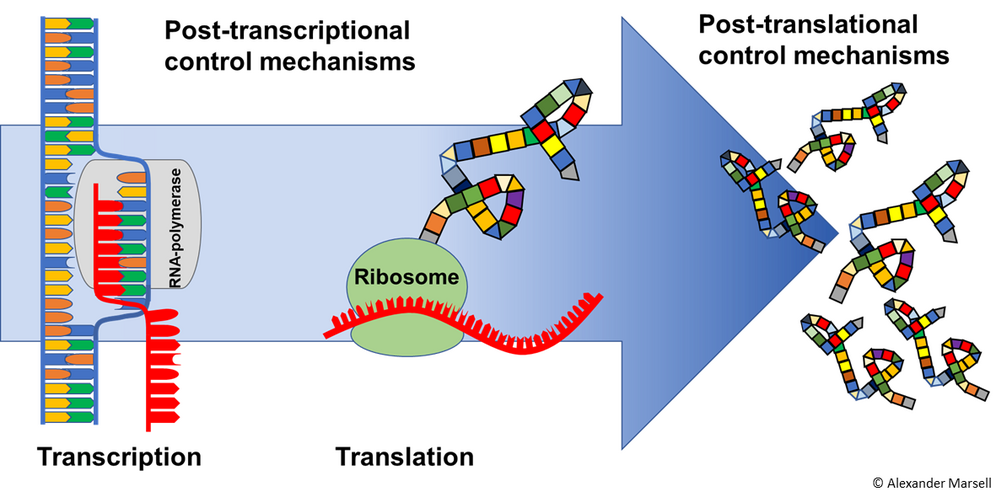
Translationskontrolle der Pathogen-regulierten Genexpression in der Arabidopsis Wurzel: genomweite und genspezifische Ansätze
Neue Studien haben gezeigt, dass die Umprogrammierung der Translation ein schneller Mechanismus ist, um auf Pathogene zu reagieren. In einer dynamischen Umwelt trägt die translationale Kontrolle der Genexpression maßgeblich zum Überleben der Pflanze bei, da sie eine Feinabstimmung der Proteinsynthese unter optimalen bzw. energiebegrenzenden Bedingungen ermöglicht. Durch die TRAPseq-Technik (Translating Ribosome Affinity Purification und anschließende RNA-Sequenzierung) haben wir die Möglichkeit, das Translatom durch Immunpräzipitation von markierten Ribosom/mRNA-Komplexen zu bestimmen. Der Vergleich von Translatom und Transkriptom zeigt nach Pathogenbefall der Wurzel nur eine geringe Korrelation. Studien nach Infektion mit Blattpathogenen kamen zu ähnlichen Ergebnissen (Xu et al., 2017), was die Bedeutung der Translationskontrolle nach Pathogenbefall hervorhebt. Ziel unseres Projektes ist es, Mechanismen zu entschlüsseln, die die Translation in der Arabidopsis Wurzel als Reaktion auf das Pathogen Verticillium longisporum anpassen.
Literatur: Fröschel et al., Cell Host and Microbe 2021
Mitarbeiter: Alexander Marsell