Pflanze-Pathogen-Interaktion
Pflanze-Pathogen-Interaktion
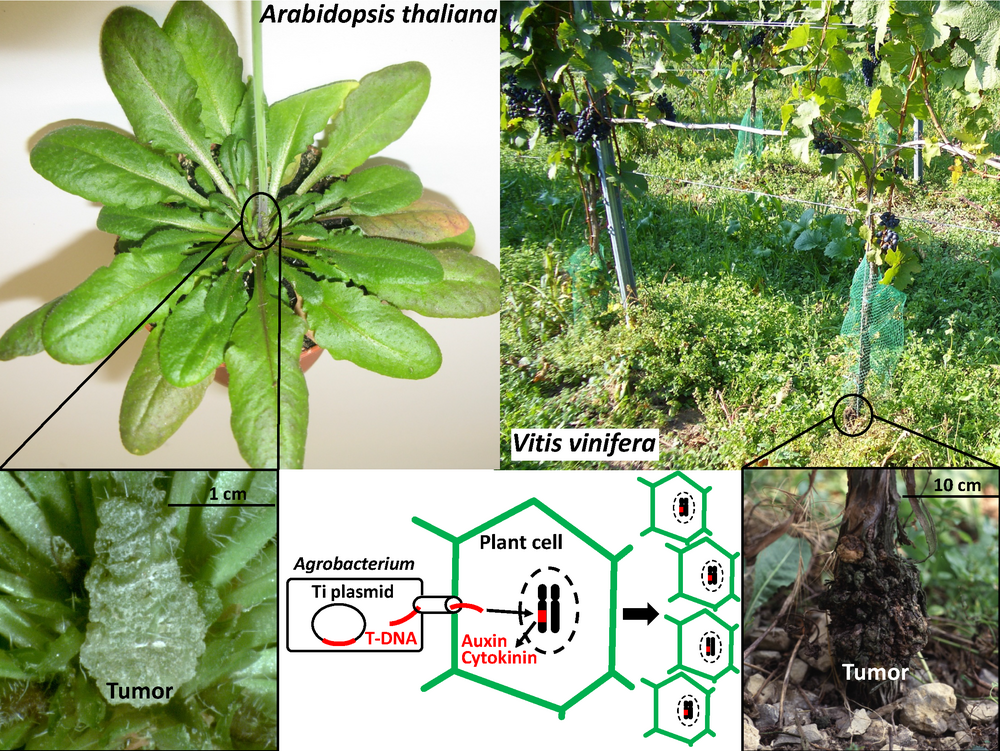
Im Fokus unserer Forschungsarbeiten stehen Wurzelhalsgallentumore, welche nach Infektion mit Agrobacterium Spezies bei Pflanzen ausgelöst werden. Das tumorauslösende Agrobakterium transformiert die Pflanzenzelle, indem es ein DNA-Fragment, die T-DNA, ins Genom der Pflanze transferiert. Aufgrund dieser Fähigkeit wird das Agrobacterium weltweit als biotechnologisches Werkzeug zur Herstellung gentechnisch veränderter Pflanzen genutzt. Die mit natürlicher T-DNA transformierten Pflanzenzellen proliferieren, was schließlich als tumorartige Wucherung sichtbar wird. Wirtschaftlich interessante Nutzpflanzen wie Weinreben, Obst- und Nussbäume, sind besonders empfänglich für Agrobakterien und die damit einhergehende Tumorbildung. Tumorisierte Pflanzen bedeuten Ernteverluste und damit einen wirtschaftlichen Schaden. Aus diesem Grunde ist es von Interesse, die Prinzipien des Transformationsprozesses und die Biologie der Tumorentwicklung zu kennen.
Mit diesem Projekt wollen wir klären, welche Signalwege der Wirtspflanze auf eine Agrobacterium-Infektion reagieren und die Entwicklung des Tumors unterstützen. Uns interessieren dabei insbesondere die molekulargenetischen Mechanismen und physiologischen Prozesse, welche die Entwicklung eines Pflanzentumors unterstützen.
In unseren Experimenten untersuchen wir:
— epigenetische Veränderungen an der DNA,
— die Regulation der Genexpression,
— die Funktionen Tumor-spezifisch exprimierter Proteine
— Änderungen im Stoffwechsel
— Genommanipulationen
— Mutanten und transgenen Pflanzen.
Anhand der Hochdurchsatztechniken wurde das Methylom, Transkriptom und Metabolom der Wurzelhalsgallen umfassend analysiert, um ein integriertes Bild von der Biologie des Tumors zu entwickeln. Unsere Studien führen wir in erster Linie an Arabidopsis thaliana durch, weil für diese Modellpflanze der Molekularbiologen eine große Vielfalt an Methoden entwickelt wurde. Aus diesem Methodenspektrum setzen wir folgende für unsere Studien ein:
— Microarrays
— Deep Sequencing
— Quantitative ‚real time‘ PCR
— Agrobacterium-vermittelte Transformationstechniken
— verschiedene Proteinexpressionssysteme
— Protein-DNA-Bindeassays
— DNA-Immunoprezipitation
— RNA Interference
— CRISPR/Cas