Das Ribosom als Kontrolleur
29.09.2016Die Synthese von makromolekularen Maschinen im Zellinneren ist ein komplizierter und diffiziler Prozess. Biochemiker aus Würzburg und Göttingen haben jetzt neue Details dieser Vorgänge entschlüsselt. Sie zeigen, dass die Zelle viel dafür tut, Produktionsfehler zu vermeiden.
Der Zusammenbau von Proteinen zu größeren makromolekularen Gebilden im Zellinneren ist an Ribosomen – und damit an ihren Entstehungsort durch den Prozess der sogenannten Translation – gekoppelt. Das ist das Ergebnis neuester Untersuchungen von Wissenschaftlern der Universität Würzburg und vom Max-Planck-Institut (MPI) für biophysikalische Chemie in Göttingen. Das Ribosom übernimmt dabei quasi die Funktion eines „Qualitäts-Checkpoints“: Es stellt sicher, dass neu gebildete Proteine direkt in die Produktionsstraße von makromolekularen Komplexen geschleust werden. Die Ergebnisse ihrer Arbeit haben die Forscher in den Fachzeitschriften Cell Reports und The EMBO Journal veröffentlicht.
Ein molekulares Legospiel
„Man muss sich das wie ein molekulares Legospiel vorstellen: Ein Baustein wird an den anderen gefügt, so lange bis das Produkt fertig ist. Kommt an einer Stelle ein fehlerhafter Stein zum Einsatz, kann am Ende das ganze Gebilde nicht funktionieren.“ Professor Utz Fischer ist Inhaber des Lehrstuhls für Biochemie an der Universität Würzburg. Schon seit vielen Jahren forscht er daran, wie sogenannte „makromolekulare Maschinen“ in Zellen zusammengebaut werden. Sein Spezialgebiet sind die Spleißosomen – große Komplexe aus Proteinen und RNA-Molekülen, die im Inneren des Zellkerns die Übertragung des genetischen Codes in Proteine kontrollieren. Sie entfernen dabei aus der Boten-RNA diejenigen Abschnitte, die keine Protein-kodierenden Informationen enthalten, und fügen die informationstragenden Abschnitte wieder zusammen.
In ihrer jüngsten Arbeit hat Fischers Team in Zusammenarbeit mit Göttinger Kollegen den Produktionsweg von sogenannten UsnRNPs, den Untereinheiten, aus denen sich die Spleißosomen zusammensetzen, komplett entschlüsselt – von der Synthese der Einzelkomponenten über deren Zusammenbau bis zur Kombination der funktionstüchtigen Maschine. Dabei haben sie einen Akteur identifiziert, dessen Rolle so bisher noch nicht bekannt war: das Ribosom.
Die Rolle der Ribosomen
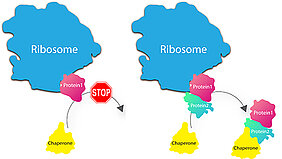
Ribosomen sind der Ort, an dem im Inneren der Zelle genetische Information in Proteine umgesetzt wird – in der Fachsprache „Translation“ genannt. Wie sich diese Proteine anschließend zu makromolekularen Maschinen zusammenfinden, war bislang nicht bis ins letzte Detail aufgeklärt. Klar war allerdings: Die Vorstellung, dass das Ribosom die Bausteine in die Zelle entlässt, wo sie so lange herumwandern, bis sie ihr passendes Gegenstück finden, konnte definitiv nicht zutreffen. „Dafür herrscht im Zellinneren ein viel zu großes Gedränge“, sagt Ashwin Chari, Projektgruppenleiter am MPI für biophysikalische Chemie. Die Proteine würden viel zu lange benötigen, um sich zu Komplexen zusammenzuschließen, da sie an „falschen“ Bestandteilen hängen bleiben und so Verklumpungen bilden, die im schlimmsten Fall gravierende Funktionsstörungen verursachen. Molecular Crowding heißt diese drangvolle Enge unter Wissenschaftlern.
„Es muss also in der lebenden Zelle einen Mechanismus geben, der die neu synthetisierten Proteine am Ribosom schützt und mit dem Wunschpartner verbindet“, sagt Elham Paknia, die experimentell das Projekt leitete. Dass dem tatsächlich so ist, konnten die Wissenschaftler jetzt erstmals nachweisen. Demnach entlässt das Ribosom die Proteine nach der Synthese nicht in das Zytosol. Stattdessen hält es sie dort so lange fest, bis bestimmte Helfer – sogenannte Chaperone – die passenden Gegenstücke anliefern. Damit stelle das Ribosom sicher, dass nur die eine, gewünschte Struktur ausgebildet werden kann; es übernimmt sozusagen neben der Produktion auch noch die Rolle eines Qualitätskontrolleurs.
Hoher Aufwand für Regulation und Kontrolle
„Extrem hohe Sicherheitsstandards“ sind nach den Erkenntnissen der Forscher sowieso ein grundlegendes Prinzip der Arbeitsweise von Zellen. So sind, wie sie zeigen konnten, an dem Zusammenbau makromolekularer Maschinen häufig mehr Helfer als Bausteine beteiligt. Das spiegelt sich auch im Energieverbrauch der Zelle wider: „Die eigentliche Katalyse verbraucht längst nicht so viel Ressourcen wie Regulation und Kontrolle. In diese Aufgaben fließt wesentlich mehr Energie hinein“, sagt Utz Fischer.
Der hohe Aufwand ist gerechtfertigt: Fehler beispielsweise beim Zusammenbau der Spleißosomen sind ein Auslöser der Spinalen Muskelatrophie. Die Krankheit ist gekennzeichnet durch ein Absterben der motorischen Nervenzellen vor allem im Rückenmark, was bei den Betroffenen zu Muskelschwund und Lähmungen führt. Fehlgefaltete Proteine gelten außerdem als Verursacher zahlreicher anderer Krankheiten – von Diabetes bis zu Alzheimer.
Ein generelles Prinzip
Auch wenn Fischer und seine Göttinger Kollegen die Rolle des Ribosoms beim Zusammenbau von Makromolekülen jetzt am Beispiel des Spleißosoms entschlüsselt haben, sind die Forscher davon überzeugt, dass es sich nicht um einen Einzelfall handelt. „Es gibt gute Gründe dafür zu glauben, dass es sich dabei um ein generelles Prinzip handelt“, sagt Fischer. Schließlich müssen auch andere Makromoleküle in der gleichen drangvollen Enge, aber mit höchsten Sicherheitsstandards synthetisiert werden.
The Ribosome Cooperates with the Assembly Chaperone pICln to Initiate Formation of snRNPs. Elham Paknia, Ashwin Chari, Holger Stark, Utz Fischer. Cell Reports, Volume 16, Issue 12, 20 September 2016. dx.doi.org/10.1016/j.celrep.2016.08.047
Reconstitution of the human U snRNP assembly machinery reveals stepwise Sm protein organization. Nils Neuenkirchen, Clemens Englbrecht, Jürgen Ohmer, Thomas Ziegenhals, Ashwin Chari & Utz Fischer, The EMBO Journal. DOI 10.15252/embj.201490350
Kontakt
Prof. Dr. Utz Fischer, T: (0931) 31-84029, E-Mail: utz.fischer@biozentrum.uni-wuerzburg.de
Dr. Ashwin Chari, T: (0551) 201-1654, E-Mail: ashwin.chari@mpibpc.mpg.de