Long-range Regulation
Biologische und klinische Relevanz von konservierten nicht-kodierenden Elementen/Long-range Regulation
Strukturelle Variabilität und daraus resultierende Konsequenzen für die phänotypische Diversität und Ausprägung von Erkrankungen sind nicht beschränkt auf die Protein-Kodierenden Bereiche unseres Genoms. Eigene Ergebnisse zeigen, dass CNVs von evolutionär konservierten nicht-kodierenden Elementen mit angeborenen Fehlbildungen assoziiert sein können. Vor allem in der direkten Nachbarschaft von entwicklungsbiologisch relevanten Genen liegen große nicht-kodierende Bereiche, die auch als Genwüsten ("gene deserts") bezeichnet werden. Es ist bekannt, dass konservierte nicht-kodierende Elemente als sogenannte long-range Regulatoren (Enhancer, Repressor) über Entfernungen von mehr als einer Megabase auf ihre Zielgene wirken können. Die präzise Kontrolle der räumlichen und zeitlichen Expression von Genen ist ein kritischer Prozess während der Embryonalentwicklung, z.B. für die Musterbildung während der Extremitätenentwicklung.
Wir interessieren uns für die zugrunde liegenden Mechanismen der long-range Regulation, d.h. wie, wo und wann interagieren die Regulatoren mit dem Zielgen. Es sollen grundlegende Fragestellungen bearbeitet werden: welche Beziehung besteht zwischen der genomischen Sequenz und ihrer funktionellen und strukturellen Konformation im Zellkern, welche Proteine/Protein-Komplexe sind an den Interaktionen beteiligt.
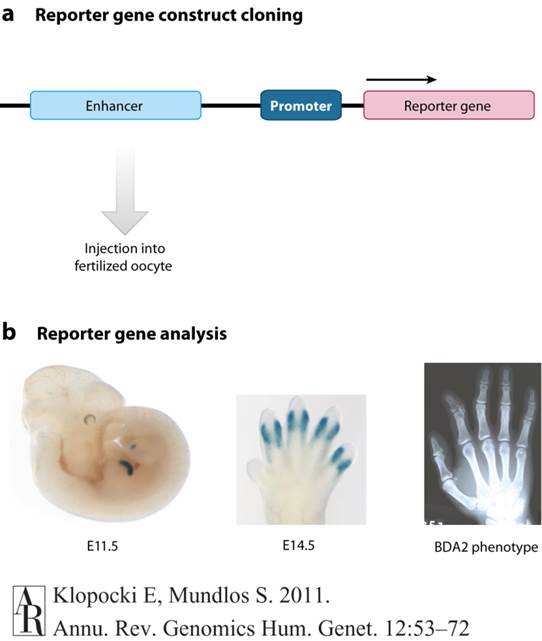
Reporter gene assay. Sequences that harbor conserved elements can be investigated for regulatory potential by in vivo reporter gene assays. (a) In the first step, the orthologous mouse sequence harboring the putative enhancer is cloned in a reporter vector upstream of a minimal promoter and a reporter gene – in this case, LacZ. Following injection of the reporter construct into fertilized oocytes, transgenic embryos are harvested at the time points of interest. (b) Analysis of reporter gene expression in these transgenic mouse embryos – in this case, by X-gal staining – reveals the ability of the putative enhancer to induce specific gene expression. In this example, a limb-specific staining at different embryonic stages (E11.5 and E14.5) that resembles the expression pattern of the target gene Bmp2 is induced by the putative enhancer – i.e., a conserved sequence 3' of Bmp2 (left and middle). Duplications of this limb-specific enhancer are associated with the BDA2 phenotype – i.e., a shortened, triangular-shaped middle phalange and radial deviation of the index finger (right) (Dathe et al. 2009).