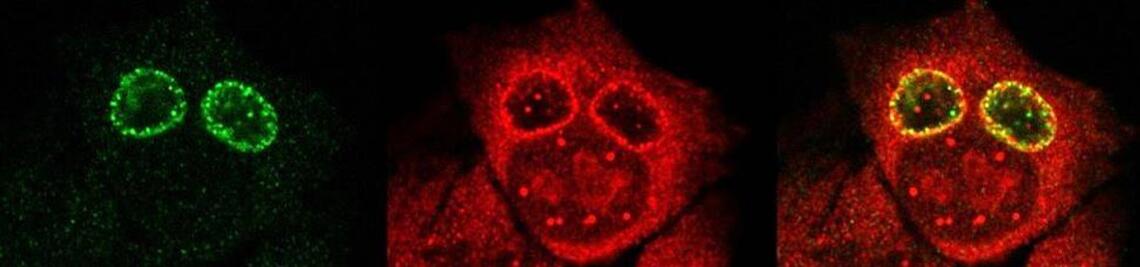
Zusammenwirkung der epithelialen Übergangszone und HPV/Chlamydien bei der Pathogenese
Epitheliale Übergangszonen (ÜZ) des Gebärmutterhalses, die Grenze, an der zylinderförmige endozervikale und squamöse ektozervikale Epithelien zusammentreffen, stellen kritische Zonen mit erhöhter Anfälligkeit für lokalisierte Infektionen mit humanen Papillomaviren (HPV) und Chlamydien dar. Die ÜZ ist häufig für eine metaplastische Anpassung prädisponiert, die Neoplasien vorausgeht, bei denen ein Epitheltyp den benachbarten ersetzt. Mehrere Faktoren wie Ernährung, saure Mikroumgebung und Hormone werden mit der Entwicklung von Metaplasien in Verbindung gebracht. Interessanterweise erhöhen aktive metaplastische Veränderungen im ÜZ des Gebärmutterhalses das Risiko einer HPV-Infektion. Darüber hinaus wird angenommen, dass Chamydia-Infektionen ein starker Kofaktor für die HPV-induzierte Pathogenese sind. Trotz dieser starken Assoziation gab es bisher keine systematische Analyse, warum die zervikale ÜZ anfälliger für Infektionen ist und wie Infektionen die ÜZ-Nische und das Stammzellverhalten während der Pathogenese beeinflussen. Hier wollen wir untersuchen, wie HPV und Chlamydien allein oder in Kombination die Nische der zervikalen ÜZ beeinflussen, einschließlich epithelialer, stromaler und immunologischer Zellkompartimente. Wir werden Chlamydieninfektionen der zervikalen ÜZ in WT- und KRT14-HPV-transgenen Mäusen etablieren, die die Gene der frühen HPV-Region exprimieren. Darüber hinaus werden wir klären, wie Chlamydien die zervikale ÜZ von WT- und HPV16-transgenen Mäusen besiedeln, wie die Ernährung die Infektionen beeinflusst und sich auf die Homöostase und Reparatur der ÜZ-Nische auswirkt. Das Ausmaß der Veränderungen in den Gewebekompartimenten wird mit modernsten Technologien wie Organoidmodellen, genetischer Abstammungsanalyse, Multiplex-Einzelmolekül-RNA-In-situ-Hybridisierung (smRNA-ISH), Multicolor-Durchflusszytometrie, Einzelzell-RNA-Sequenzierung (scRNA) und räumlicher Transkriptomik untersucht.