Genexpressionskontrolle durch regulatorische RNAs in Neisseria gonorrhoeae
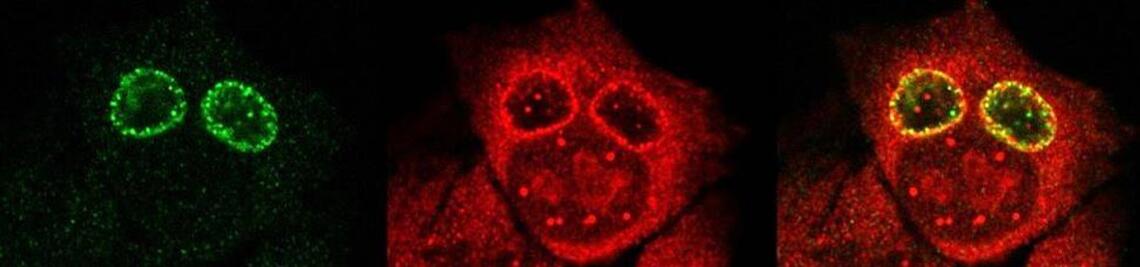
Nicht-codierende RNAs kontrollieren die Genexpression auf post-transkriptioneller Ebene und sind wichtige regulatorische Elemente in der bakteriellen Stressantwort und Pathogenität. Kleine regulatorische RNAs (sRNAs) weisen üblicherweise eine Länge von 50 bis 500 Nucleotiden auf und werden von intergenischen Regionen des bakteriellen Genoms transkribiert. Diese sRNAs wirken in trans und zeigen nur begrenzte Komplementarität mit ihren Ziel-mRNAs. Sie binden meist überlappend oder in unmittelbarer Nähe der Ribosomenbindestelle auf der mRNA und beeinflussen so deren Translation (Abb. 1).
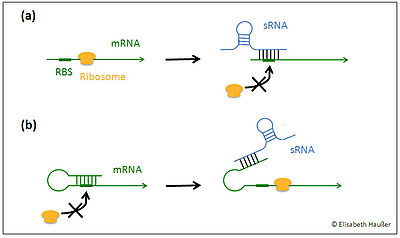
Abb. 1: Mechanismen der post-transkriptionellen Genregulation durch bakterielle sRNAs:
(a) Inhibition der Translationsinitiation: Hybridisierung der asRNA mit der 5‘-UTR der mRNA wird die Ribosomenbindestelle maskiert und somit die Initiation der Translation unterbunden.
(b) Aktivierung der Translation: Durch Hybridisierung mit einem Bereich der 5‘-UTR der mRNA wird eine inhibitorische Sekundärstruktur der mRNA aufgelöst und somit die Ribosomenbindestelle für das Ribosom zugänglich.
Die Ausbildung von mRNA-sRNA Komplexen kann zudem Auswirkungen auf die Stabilität der mRNA haben. Antisense-RNAs (asRNAs) werden vom Gegenstrang einer codierenden Region transkribiert und zeigen ausgeprägte Heterogeniät bezüglich ihrer Länge, die von weniger als hundert bis zu mehreren Tausend Nucleotiden reichen kann. Ihre Funktion ist weniger gut verstanden, es könnte jedoch bereits gezeigt werden, dass asRNAs mRNA-Stabilität, Translationseffizienz und Transkriptions-Termination modulieren oder Genexpression durch transkriptionelle Interferenz beeinflussen können. Die RNA-seq basierte Transkriptomanalyse des humanpathogenen Bakteriums Neisseria gonorrhoeae deutete auf ein beträchtliches Repertoire an nicht-codierenden, regulatorischen RNAs hin (Remmele et al., 2014). Bei 59 Transkripten könnte es sich gemäß ihrer Genomlokalisation um regulatorische sRNAs handeln. Für 194 Gene konnten asRNAs detektiert werden, wobei es sich entweder um nicht-codierende Transkripte handelt, die sich von der codierenden Region des auf dem Gegenstrang lokalisierten Gens ableiten, oder aber um lange 5‘- oder 3‘ nicht-translatierte Bereiche von Protein-codierenden mRNAs, die mit mRNAs von Genen mit entgegengesetzter Transkriptionsrichtung überlappen. Transkripte, die mit dem RNA-Chaperon Hfq interagieren, das häufig an der sRNA-vermittelten Expressionskontrolle beteiligt ist, konnten bereits identifiziert werden. Ziel dieses Projekts ist die funktionelle Charakterisierung ausgewählter sRNAs und asRNAs von N. gonorrhoeae. Interessanterweise, treten in N. gonorrhoeae asRNAs häufig assoziiert mit Genen auf, die aufgrund der Anwesenheit kurzer repetitiver Sequenzmotive in ihrer codierenden Region einer Phasenvariation unterliegen. In diesem Zusammenhang soll untersucht werden, ob asRNAs die Halbwertszeit von mRNAs, die sich von phasenvariablen Genen ableiten, in Abhängigkeit des Leserahmens modulieren können.